Künstliche Intelligenz in Rhinodiagnost
Problemstellung: Segmentierung (Erzeugung von inhaltlich zusammenhängenden Regionen, hier: Bestimmung von Nasenhöhlen und Nasennebenhöhlen aus CT-Bildern) mittels eines „Convolutional Neural Networks“ (CNN) unter Einsatz von Google-TensorFlow
Damit aus Bilddaten ein 3D Modell erzeugt werden kann, sind jene Bildpunkte zu selektieren, die das 3D Modell darstellen soll. Doch CT Aufnahmen haben ein gewisses Rauschen (Störungen, die keinen Bezug zum eigentlichen Bildinhalt haben), da die Radioquelle zum Wohle des Patienten mit geringstmöglicher Intensität betrieben wird, und manche Strukturen in den CT Bildern sind so fein, dass sie in der Auflösung nicht mehr korrekt erfasst werden. Daher müssen die Bilder für die Erzeugung des 3D Modells jedenfalls nachbearbeitet werden. Im vorliegenden Versuch wurde der manuelle Nachbearbeitungsprozess durch Machine Learning und Convolutional Neural Networks ersetzt. Als Trainingssatz wurden 160 Bilder (Axialschnitte) einer Person ausgewählt und in den Algorithmus eingespeist.
Die Bildreihe in Abbildung 1 zeigt beispielhaft fünf CT Bilder, die als Ausgangsdaten dienten.
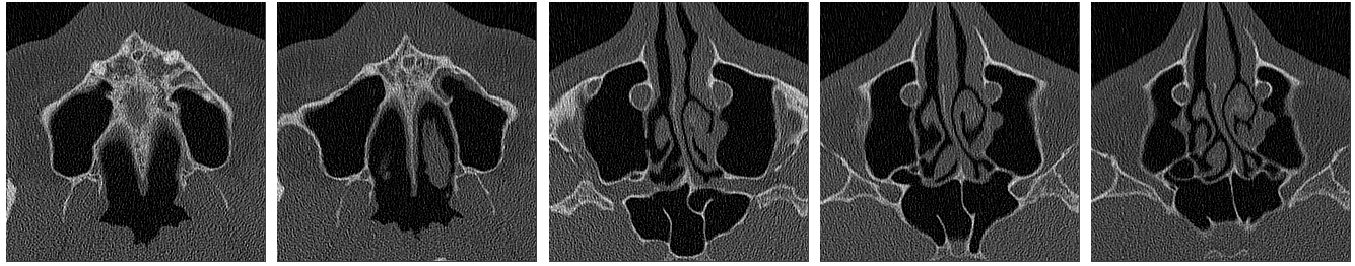
Abbildung 1 Testdaten (5 dem CNN bislang nicht bekannte CT-Schnittbilder)
Bei der Segmentierung werden im Bild inhaltlich zusammenhängende Regionen erzeugt. Die Bildreihe in Abbildung 2 zeigt die vom CNN berechneten Wahrscheinlichkeiten, ob ein Bildpunkt Teil der Segmentierung ist. Die Bilder werden mit kontinuierlichen Farbwerten gezeigt.
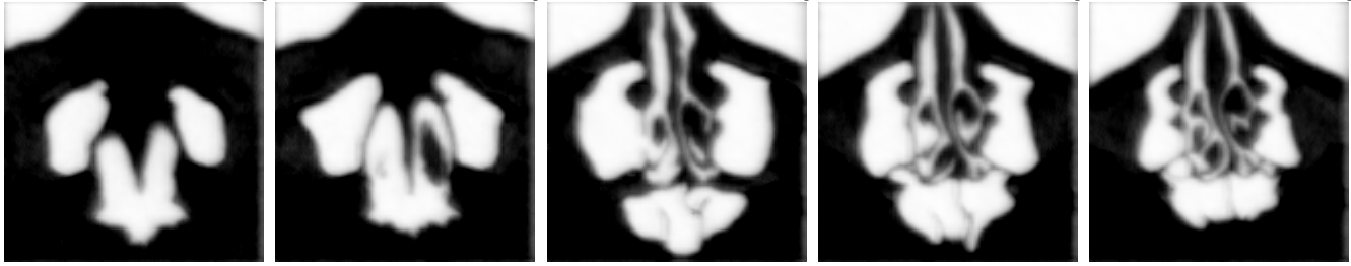
Abbildung 2 Vom CNN vorhergesagte Farbwerte
Anschließend wurde vom CNN eine binäre Segmentierung, Reduktion auf die Farbwerte Weiß und Schwarz, generiert. („binär“: weiß=im Hohlraum, schwarz=außerhalb einer Nasenhöhle). Diese Segmentierung ist in Abbildung 3 dargestellt.

Abbildung 3 Vom CNN generierte Segmentierung (binär)
In Abbildung 4 sieht man die von Hand erstellten Segmentierungen, wobei hier ebenfalls nur noch die Farbwerte Weiß und Schwarz verwendet wurden.
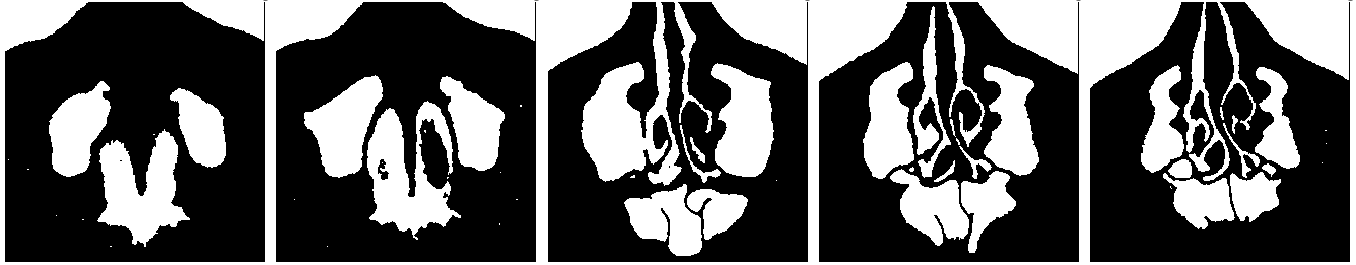
Abbildung 4 Von Hand erstellte Segmentierung (binär)
Um die manuelle Segmentierung bestmöglich zu simulieren, musste nun der Übergang von der zweiten zur dritten Bildreihe, die Reduktion auf die zwei Farbwerte Weiß und Schwarz, verbessert und optimiert werden. In einem weiteren Versuch wurden die Parameter des neuronalen Netzes (Schichttiefe und Lernrate) verändert, wodurch ein deutlich verbessertes Ergebnis erzielt wurde (Abbildung 5).
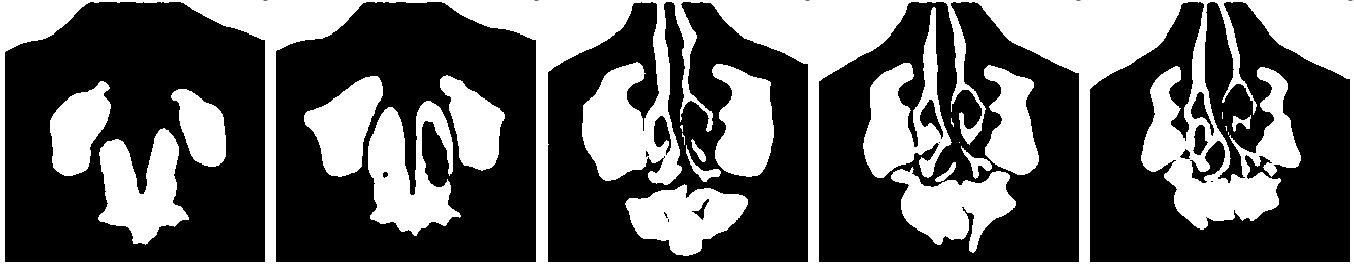
Abbildung 5 Vom CNN erstellte Segmentierung mit veränderten Parametern
Wesentlich bei diesem ersten Versuch war die Überprüfung, ob die gewählte Methode (CNN) zu einem brauchbaren Ergebnis führt, wobei es das Ziel war, zu erkennen ob ein Punkt des CT-Scans (DICOM-Image) innerhalb oder außerhalb einer Nasen(neben)höhle liegt. Obwohl nur wenige Bilder einer Person für den Trainingssatz verwendet wurden, ergab sich trotz des geringen Datenmaterials eine der manuellen Bearbeitung ähnliche, oder sogar verbesserte, Segmentierung wie ein Vergleich der Bildreihen in den Abbildungen 4 und 5 zeigt.
In der Folge wird das CNN nun durch Einbeziehung größerer Datenmengen (ca.1500 Personen, je ca. 250 Bilder) und die Optimierung der Parameter weiter verbessert. Schlussendlich soll die Entwicklung des neuronalen Netzes zu einer Klassifizierung führen die angibt in welchem Zustand sich eine Nasen(neben)höhle befindet (Lage des krankhaften Gewebes, etc.) und die es ermöglicht, dass die zugehörige 3D-Darstellung (STL-File) automatisch erzeugt werden kann. Damit wird die Grundlage zur weiteren Aufbereitung und Untersuchung einer 3D-modellierten Region (RoI, Region of Interest) wie 3D-Druck, CFD-Simulation, Virtual Surgery, etc. geschaffen.
Trainingssatz: 160 Bilder/Axialschnitt, Testsatz: 10 Bilder, 5 davon sind in den Abbildungen gezeigt.
Testmaschine: Intel Core i7-7820X, 3.60 GHz; 64GB memory; 2 GPUs: GeForce GTX 1080 Ti.
Autoren: AIT Angewandte Informationstechnik Forschungsgesellschaft, Matthias Lehner, Odo Benda
